這篇主要討論各種其他技巧,但不一定有效,我們得自己實驗看看
也是提供一些不同的思路
在這一篇中,除了之前的目標,識別病的種類外,還要識別稻谷的種類。
dls = DataBlock(
blocks=(ImageBlock, CategoryBlock, CategoryBlock),
n_inp=1, #指定了輸入數據的數量。在這種情況下,我們只有一個輸入,即圖像,因此其他兩個變數(病害和品種)將被視為輸出。
get_items=get_image_files, #get數據集中的圖像文件的列表。return所有圖像文件的路徑。
get_y=[parent_label, get_variety], #使用get_y來指定兩個目標:parent_label函數將用於獲取病害類別,而get_variety函數將用於獲取水稻品種。
splitter=RandomSplitter(0.2, seed=42),
item_tfms=Resize(192, method='squish'),
batch_tfms=aug_transforms(size=128, min_scale=0.75) #這行設定了批次的變換。我們對批次中的圖像應用了一些增強變換,包括將大小調整為128x128像素,並在0.75到1之間的比例上進行縮放。
).dataloaders(trn_path)
這段程式碼的作用是創建一個DataLoaders對象,它包含了訓練和驗證數據。我們指定了數據的結構,包括圖像(ImageBlock)和兩個分類目標(CategoryBlock),分別是病害和水稻品種。我們還指定了數據的處理方式,包括圖像的變換和增強。
最後,我們使用dataloaders(trn_path)方法創建DataLoaders物件。這個物件包含了我們用於訓練和驗證模型的數據。
def disease_err(inp, disease, variety): return error_rate(inp, disease)
def disease_loss(inp, disease, variety): return F.cross_entropy(inp, disease)
這兩個函數的作用如下:
disease_err 函數計算疾病預測的錯誤率。它接收三個參數:模型的輸出 inp、疾病目標 disease 和品種目標 variety。然而,它僅使用模型的輸出和疾病目標來計算錯誤率。
disease_loss 函數計算疾病預測的損失。它同樣接收三個參數:模型的輸出 inp、疾病目標 disease 和品種目標 variety。但它僅使用模型的輸出和疾病目標來計算交叉熵損失。
這樣,我們可以在訓練過程中使用這兩個函數來評估疾病預測的性能,同時考慮兩個目標:疾病和品種。這些變化允許我們使用相同的模型架構來處理多個目標。
arch = 'convnext_small_in22k'
learn = vision_learner(dls, arch, loss_func=disease_loss, metrics=disease_err, n_out=10).to_fp16()
lr = 0.01
這邊定義了learner,使用我們定義的疾病損失函數 disease_loss 和評估指標 disease_err,並指定 n_out=10 以確保模型有 10 個輸出節點,每個節點對應一種可能的疾病
接下來我們將使用 learn.fine_tune(5, lr) 開始訓練這個模型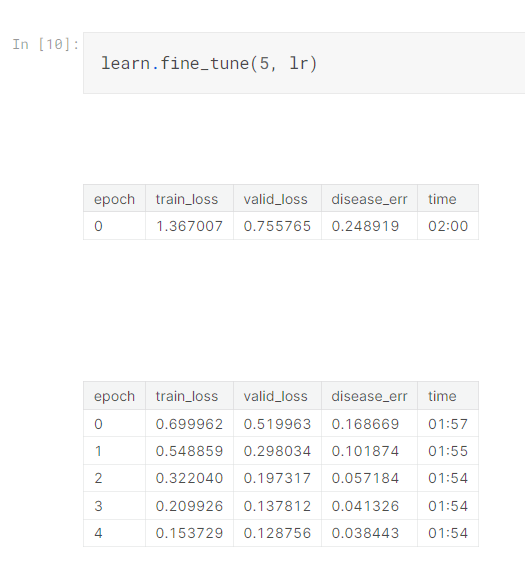
為了同時預測每種疾病和每種品種的機率,我們現在需要讓模型輸出一個長度為 20 的張量,因為有 10 種可能的疾病和 10 種可能的品種。我們可以通過設置 n_out=20 來實現這一點:
learn = vision_learner(dls, arch, n_out=20).to_fp16()
這邊的20個輸出節點,這些節點對應 10 種可能的疾病和 10 種可能的品種
接下來,我們可以像之前一樣定義 disease_loss,但有一個重要的改變:
輸入張量現在的長度為 20,而不是 10,因此不匹配可能的疾病數量。我們可以選擇要用來預測疾病的輸入部分。
我們使用前 10 個值:
def disease_loss(inp, disease, variety):
return F.cross_entropy(inp[:,:10], disease)
這意味著我們可以對預測品種做相同的事情,但使用輸入的最後 10 個值,並且用品種取代disease
def variety_loss(inp,disease,variety): return F.cross_entropy(inp[:,10:],variety)
這邊寫了一個組合的loss ,就是disease跟viariety 的加總
def combine_loss(inp,disease,variety): return disease_loss(inp,disease,variety)+variety_loss(inp,disease,variety)
為了查看這2種的error_rate ,分別寫了2個function 來查看
def disease_err(inp,disease,variety): return error_rate(inp[:,:10],disease)
def variety_err(inp,disease,variety): return error_rate(inp[:,10:],variety)
err_metrics = (disease_err,variety_err)
all_metrics = err_metrics+(disease_loss,variety_loss)
最後就把這些輸入當成vision_learner 的輸入
最後跑5次看看結果
learn = vision_learner(dls, arch, loss_func=combine_loss, metrics=all_metrics, n_out=20).to_fp16()
learn.fine_tune(5, lr)
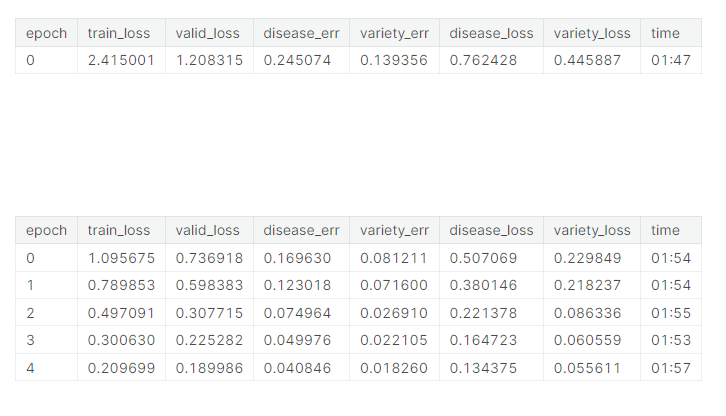
以上就結束了,講師的意思是說,這邊提供了一個多目標的辨識,但他不一定能改善單目標的辨識。
有可能有幫助,也有可能沒幫助,都可以試試看! 也是一種思路。
